Needleman-Wunsch algorithm(니들만-브니쉬(분쉬) 알고리즘)
Needleman-Wunsch 알고리즘
- 생물정보학 에서 단백질 또는 뉴클레오티드 서열 을 정렬 하는 데 사용되는 알고리즘
- 생물학적 시퀀스를 비교하기 위한 동적 프로그래밍 의 첫 번째 응용 프로그램 중 하나
- Needleman-Wunsch 알고리즘은 특히 전역 정렬의 품질이 가장 중요한 경우 최적의 전역 정렬에 여전히 널리 사용
- 알고리즘은 가능한 모든 정렬에 점수를 할당하고 알고리즘의 목적은 가장 높은 점수를 가진 가능한 모든 정렬을 찾는 것
Needleman-Wunsch 알고리즘 방법
match, mis-match, gap에 대해 score를 매김
아래 두가지의 순서를 가진 서열이 있다고 가정하면
- seq1. AGTCG
- seq2. ATGG
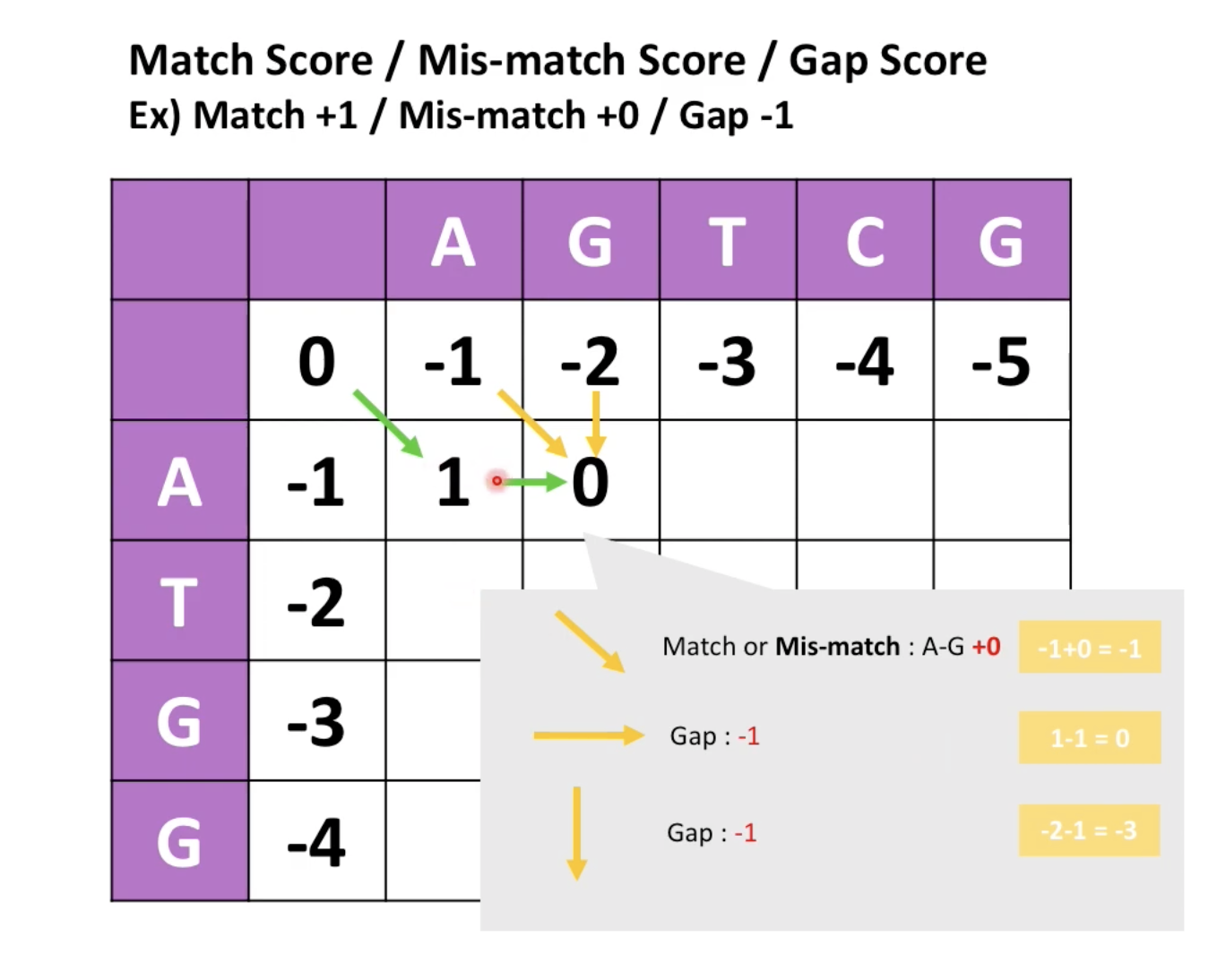
1. 두 서열을 기준으로 matrix를 만든다.
2. 정해진 규칙에 따라 매트릭스 값을 채운다.
- 각 방향별 점수를 매기고, 방향별 매겨진 점수 중 가장 큰 값을 기재
- 이때 어느방향값이 제일 큰지에 대한 정보도 남겨야함

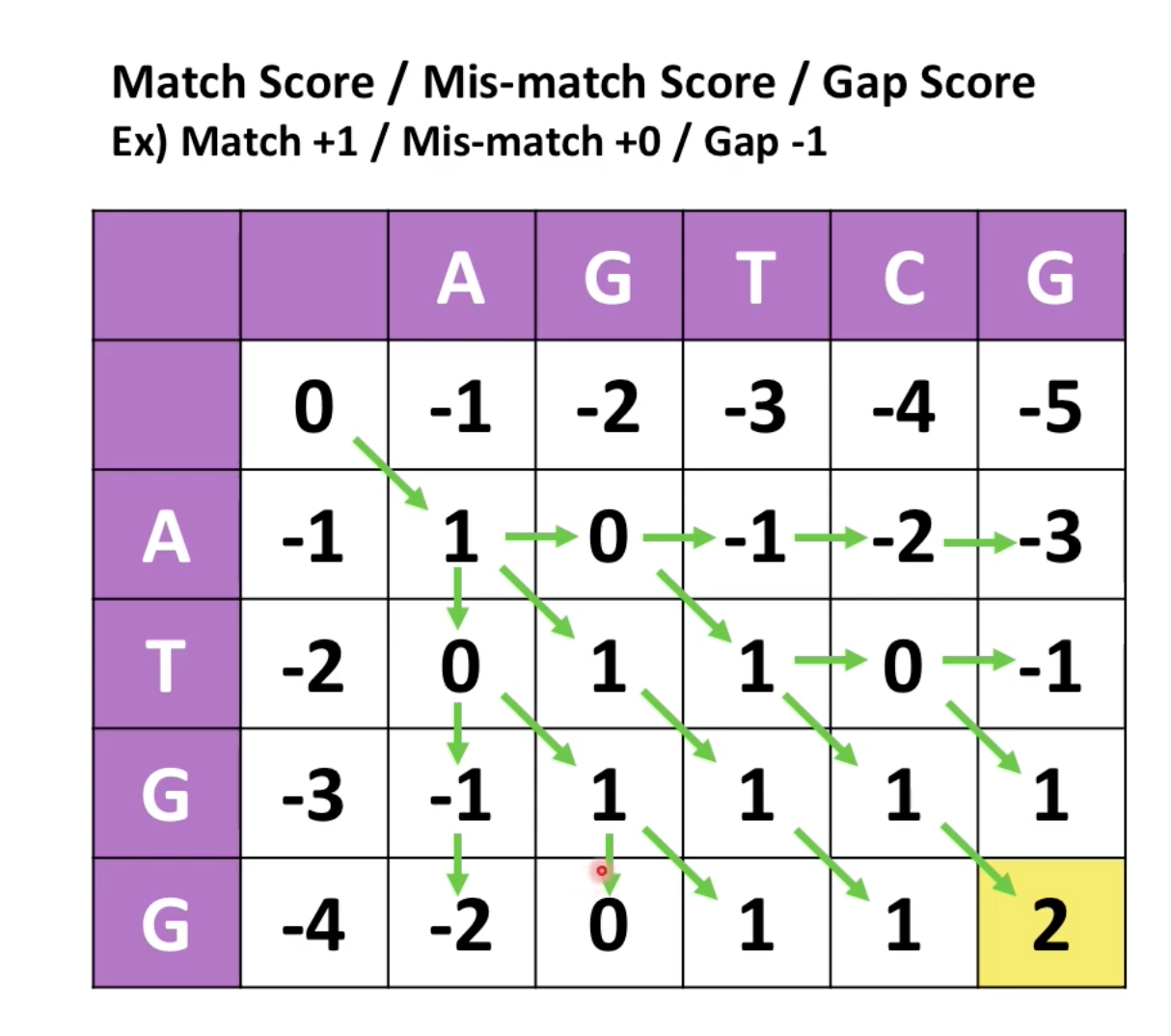
3. 매트릭스를 다 채웠으면, 오른쪽 아래 맨끝에 부터 score를 받은 방향으로 역순으로 가며 값들을 가져온다.
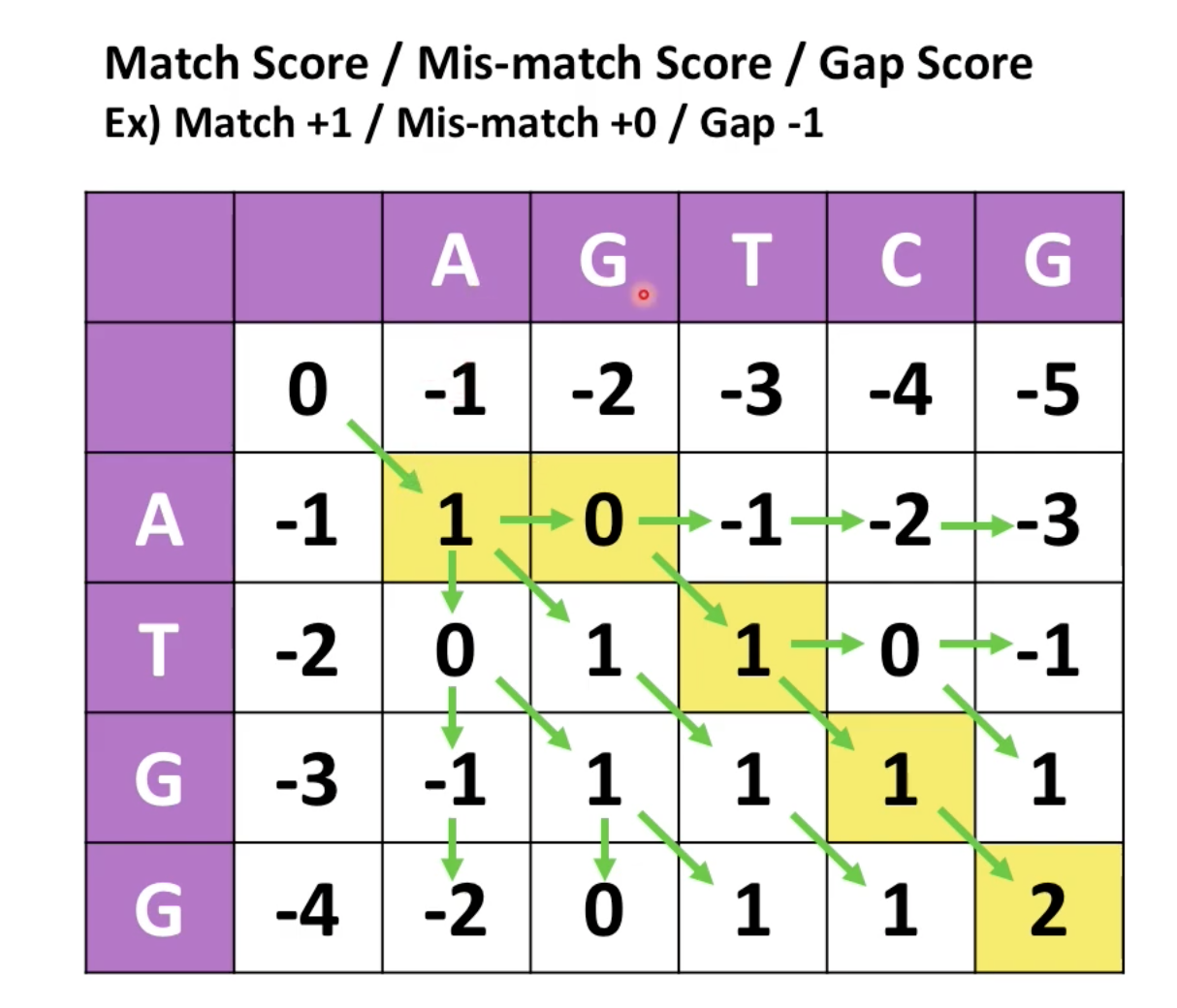
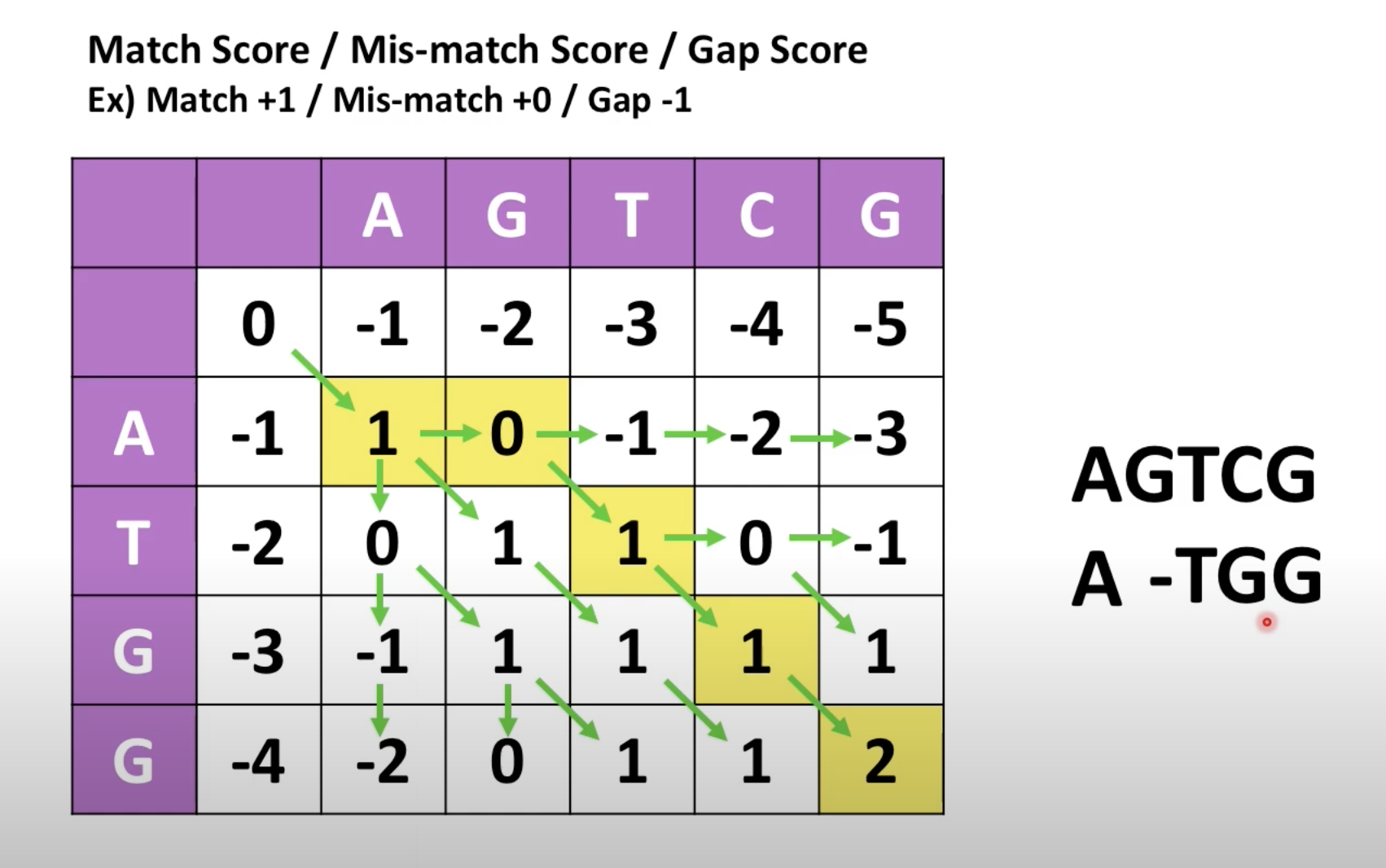
4. 최종 시퀀스 재배열을 만든다.
- 이때, A의 경우, 1과 0의 두개 값을 가지는데 즉, 같은 값이면서 gap도 가진다는 의미임
> A -(갭)TGG로 다시 나타낼 수 있음
>> 즉 AGTCG를 이용하여 ATGG가 A-TGG로 재배열을 시키는 과정이 Needleman-Wunsch 알고리즘이다.
- 유사도 시퀀스 계산
만약 니들만 분쉬 알고리즘을 이용해 아래와 같은 매트릭스를 만들었다면,
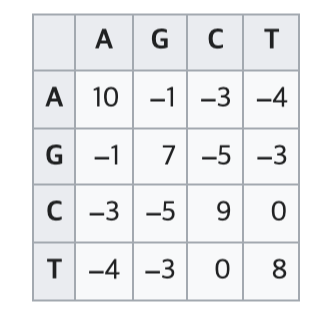
이 아래 두개의 유사도를 위 매트릭스를 기준으로 계산할 수 있다.
AGACTAGTTAC
CGA---GACGTS(A,C) + S(G,G) + S(A,A) + (3 × d) + S(G,G) + S(T,A) + S(T,C) + S(A,G) + S(C,T)
= −3 + 7 + 10 − (3 × 5) + 7 + (−4) + 0 + (−1) + 0 = 1
score가 클수록 유사도가 높음을 의미
또한 재배열된 시퀀스를 이용하여 seq1 과 seq2간의 유사도는 자카드 계수나 단순히 일치/불일치 갯수를 이용하여 유사도 계산할 수 있음
참고
Needleman–Wunsch algorithm - Wikipedia
From Wikipedia, the free encyclopedia Jump to navigation Jump to search Method for aligning biological sequences The Needleman–Wunsch algorithm is an algorithm used in bioinformatics to align protein or nucleotide sequences. It was one of the first appli
en.wikipedia.org